Chainerで癌の良性・悪性の分類予測を試してみたいと思います。
Chainerとは
Chainerは、Preferred Networks社が開発を進めているディープラーニング(ニューラルネットワーク)に特化したフレームワークです。
ディープラーニングについては以下の記事を見て頂けると嬉しいです。
case-k.hatenablog.com
Chainerのメリット
ChainerのメリットとしてはDefine by Runと呼ばれる仕組みがあります。TensorFlowや他のディープラーニングに特化したフレームワークと比較して
学習途中に数値やサイズの確認が出来るためデバックがしやすいです。また、Chainerの構成はシンプルなのでパラメータのチューニングも理解しやすいのがメリットです。
Chainer構造理解
Chainerの構成要素は以下となります。詳細については実装編で説明させて頂きますが簡単に全体像を把握できるように内容を記載します。
実装編
それでは実際にChainerで癌の分類予測問題を解きたいと思います。まずは必要となるライブラリをインポートします。
# library from sklearn.datasets import load_breast_cancer import pandas as pd import numpy as np from matplotlib import pyplot as plt %matplotlib inline import chainer import chainer.links as L import chainer.functions as F from chainer import training from chainer.training import extensions import json
cancerデータを取得しどのようなデータ確認します。
cancer = load_breast_cancer() print("cancer kesy:{}".format(cancer.keys())) print("cancer target_names:{}".format(cancer.target_names)) print("cancer target:{}".format(np.unique(cancer.target))) print("cancer feasture_names:{}".format(np.unique(cancer.feature_names))) print("cancer feasture_names shape:{}".format(np.unique(cancer.feature_names.shape))) # Out cancer kesy:dict_keys(['DESCR', 'target_names', 'target', 'feature_names', 'data']) cancer target_names:['malignant' 'benign'] cancer target:[0 1] cancer feasture_names:['area error' 'compactness error' 'concave points error' 'concavity error' 'fractal dimension error' 'mean area' 'mean compactness' 'mean concave points' 'mean concavity' 'mean fractal dimension' 'mean perimeter' 'mean radius' 'mean smoothness' 'mean symmetry' 'mean texture' 'perimeter error' 'radius error' 'smoothness error' 'symmetry error' 'texture error' 'worst area' 'worst compactness' 'worst concave points' 'worst concavity' 'worst fractal dimension' 'worst perimeter' 'worst radius' 'worst smoothness' 'worst symmetry' 'worst texture'] cancer feasture_names shape:[30]
2クラス分類問題で、入力層は30となります。扱いやすいよう、DataFrameにします。
# function def get_target_names(x): if x == 0: return "malignant" if x == 1: return "benign" cancer_data = pd.DataFrame(columns=cancer['feature_names'],data = cancer['data']) cancer_data['target'] = cancer['target'] cancer_data["target_names"] = cancer_data['target'].apply(lambda x : get_target_names(x)) cancer_data.head(5)
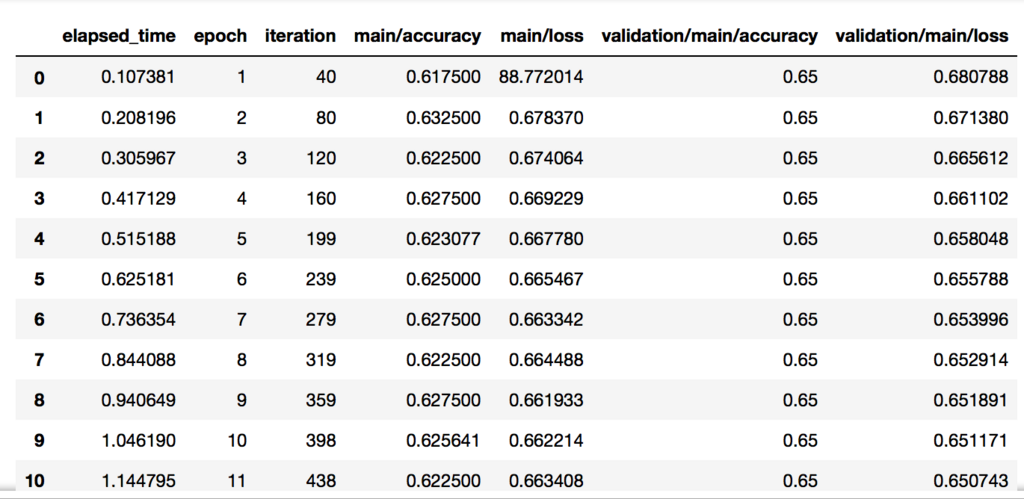
データを確認してみます。
print(cancer_data.shape) # Out (569, 32)
32カラムで569レコードのデータセットができました。入力変数と教師データ(出力変数)に切り分けます。
# 教師データ t = cancer_data.iloc[:,-2] x = cancer_data.iloc[:,0:-2] print(t.shape) print(x.shape) # Out (569,) (569, 30)
入力変数と教師データに切り分けることができました。
しかし、このままではChainerで学習させることはできません。Chainerで計算できるデータ形式に変換する必要があります。
Chainerで計算できるデータ形式に変換
Chainerで計算を行うために、下記の3点を満たしている必要があります。
- 入力変数や教師データがNumpyで定義されているか
- 分類の場合、ラベルが0から始まっているか
- 入力変数が float32、教師データが回帰の場合 float32、分類の場合 int32 で定義されているか
作ったデータが上記のルールに従っているか確かめmす。
print("type check:{}".format(type(t))) print("type check:{}".format(type(x))) print("label check:{}".format(np.unique(t.values))) print("dtype check:{}".format(t.dtype)) print("dtype check:{}".format(x.values.dtype)) # Out type check:<class 'pandas.core.series.Series'> type check:<class 'pandas.core.frame.DataFrame'> label check:[0 1] dtype check:int64 dtype check:float64
データ形式や要素のデータ型がChainerで計算できるデータ形式に
なっていないため、変換する必要があります。
x = np.array(x.astype('float32')) t = np.array(t.astype('int32')) print("type check:{}".format(type(t))) print("type check:{}".format(type(x))) print("label check:{}".format(np.unique(t))) print("dtype check:{}".format(t.dtype)) print("dtype check:{}".format(x.dtype)) # Out type check:<class 'numpy.ndarray'> type check:<class 'numpy.ndarray'> label check:[0 1] dtype check:int32 dtype check:float32
正しくデータ形式を変換させることができました。
Chainerで使用するデータセットの形式
メモリに乗の小さなデータの場合は、入力変数と教師データをタプルで1セットにし、リスト化しておくことがChainer推奨のデータ形式となります。
dataset = list(zip(x, t))
次に教師データと検証データに分割したいと思います。
# 訓練データのサンプル数 n_train = int(len(dataset) * 0.7) # 訓練データ(train)と検証データ(test)に分割 train, test = chainer.datasets.split_dataset_random(dataset, n_train, seed=1)
以上でChainerで必要とされるデータ形式が完成しました。
ここからは、modelの宣言、optimizerを定義、iteratorを定義、updaterを定義していきます。
まずはモデルの定義です。
モデルの定義
ここでは入力層・隠れ層・出力層を定義し損失関数を求めています。
詳しくは以下をご確認下さい。
case-k.hatenablog.com
# ニューラルネットワークのモデルを定義 class NN(chainer.Chain): # モデルの構造 def __init__(self, n_mid_units=5, n_out=2): super().__init__() with self.init_scope(): self.fc1 = L.Linear(None, n_mid_units) self.fc2 = L.Linear(None, n_out) # 順伝播 def __call__(self, x): u1 = self.fc1(x) z1 = F.relu(u1) u2 = self.fc2(z1) return u2
モデルを定義できたのでインスタンス化します。
np.random.seed(1) # インスタンス化 nn = NN() model = L.Classifier(nn)
これでmodelの定義とインスタンス化は完了です。
モデルの定義
次は「optimizer」を定義します。
Optimizerの定義
optimizerとはパラメータの更新を行う箇所で、パラメータの最適化を行うための最適化のアルゴリズムを選択します。
optimizer = chainer.optimizers.SGD() optimizer.setup(model)
今回は 確率的勾配降下法(SGD)を使用しました。
確率的勾配降下法については別途記事を書きたいと思います。
アルゴリズムを定義するだけでは、モデルに反映されないため
optimizer.setup(model)でアルゴリズムをモデルに適用させます。
次はIteratorを定義する必要があります。
Iteratorの定義
Iteratorでは「バッチサイズ」を決めます。順伝播で評価関数を計算するとき全てのサンプルを使用するのではなく、ミニバッチと呼ばれるサンプルの一部のデータセットのみで評価関数の計算を行いパラメータの学習を行います。
バッチサイズとは
「バッチサイズ」とはランダムに抽出したサンプルの一部のデータセットのことです。
100万レコードある場合、100万レコードいっぺんに実施することはメモリやCPUが足りません。
なので100サンプルずつ実施するようにします。この1回の試行で利用するデータのサイズをバッチサイズといいます。
またバッチサイズ1回行うことを1 epochといいます。
100万レコードを、100サンプルずつ行う場合1万epochとなります。
batchsize = 10 train_iter = chainer.iterators.SerialIterator(train, batchsize) test_iter = chainer.iterators.SerialIterator(test, batchsize, repeat=False, shuffle=False)
Iteratorを定義できました。次はupdaterの定義です。
updaterの定義
Updaterでは、Optimizerの設定・デバイス(CPUやGPU)の設定を行うことができます。
CPUを利用する場合、device=-1とオプションに指定し、GPUを使用する場合にはdevice=0
と明示しておく必要があります。deviceを指定しない場合は、CPUが使用されます。
今回はCPUを利用します。
updater = training.StandardUpdater(train_iter, optimizer, device=-1)
updaterを定義できました。次はTrainerとextensionsの設定を行います。
Trainerとextensionsの設定
Trainerでは、エポック(ミニバッチを全て処理して1エポック)の回数や、
そのextensionsでオプションを指定することにより、結果をログ出力や標準出力できます。
# エポックの数 epoch = 50 # trainerの宣言 trainer = training.Trainer(updater, (epoch, 'epoch'), out='result/cancer') # 検証データで評価 trainer.extend(extensions.Evaluator(test_iter, model, device=-1)) # 学習の経過をtrainerのoutで指定したフォルダにlogというファイル名で記録する trainer.extend(extensions.LogReport(trigger=(1, 'epoch'))) # 1エポックごと(trigger)に、trainデータに対するlossと、testデータに対するloss、経過時間(elapsed_time)を標準出力させる trainer.extend(extensions.PrintReport(['epoch', 'main/accuracy', 'validation/main/accuracy', 'main/loss', 'validation/main/loss', 'elapsed_time']), trigger=(1, 'epoch')) # 実行 trainer.run()
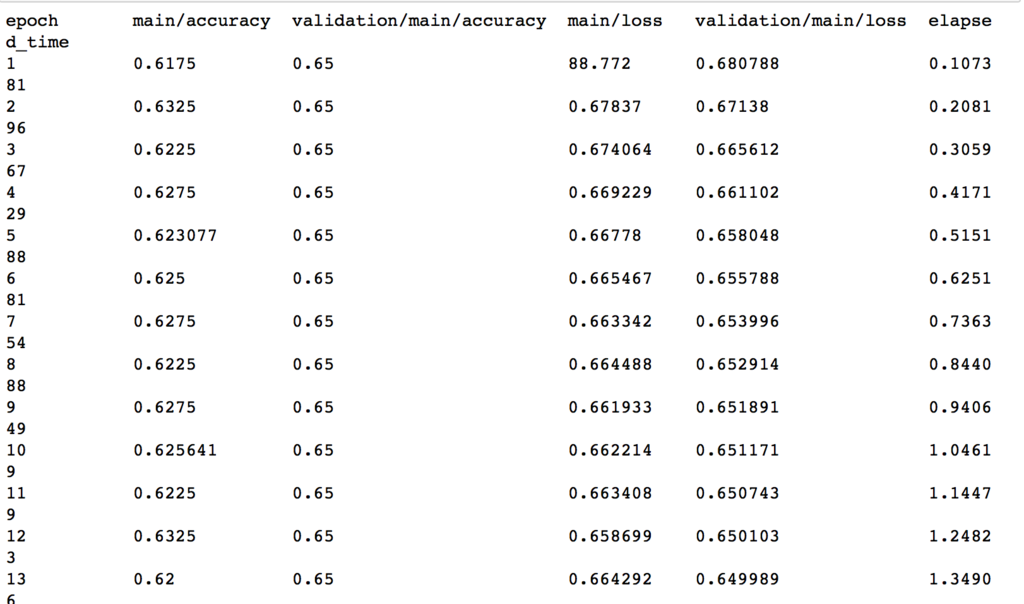
実行結果を確認してみてみましょう。Trainerを使用すると、事前に定義しておいたresult/cancerというディレクトリができ、その中のlogというファイルが生成されます。
logのファイルを確認してみてみましょう。
with open('result/cancer/log') as f: logs = json.load(f) results = pd.DataFrame(logs) # 結果の確認 results
# accuracy(精度)を表示 results[['main/accuracy', 'validation/main/accuracy']].plot()
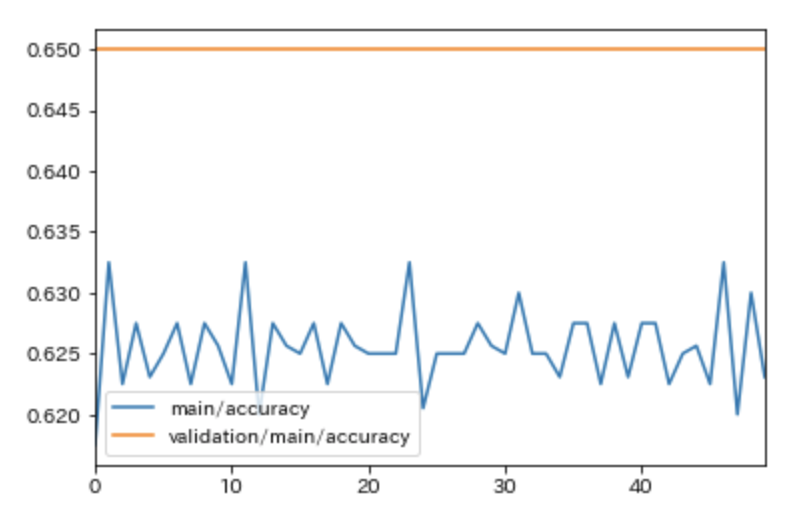
精度は訓練データに対しては62%ほどであり、
検証データに対して65%ほどであることが分かります。
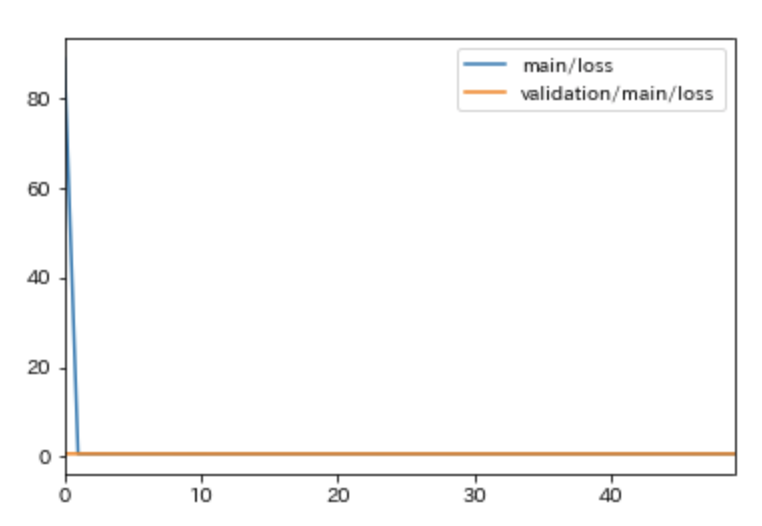
損失関数も確認してみたいと思います。
# loss(損失関数)を表示 results[['main/loss', 'validation/main/loss']].plot()
とりあえず一連の流れは理解できました。
正直色々なアルゴリズムが多く何が最適なのか判断が難しいですね。調べたところ画像認識ではChainerが優れているようです。
次はChainerで精度を向上させる方法や予測問題・他のフレームワークとも比較してみたいですね。
# 追記
ランダムフォレストでも試してみました。